Transkription
[Transkription][Transkription] w [von latein. transcriptio = Umschrift; Verb transkribieren], RNA-Synthese, RNS-Synthese, der 1. Schritt der Genexpression, bei dem es, von bestimmten RNA-Viren abgesehen, zu einer DNA-abhängigen (Desoxyribonucleinsäure fungiert als Matrize) Synthese der Ribonucleinsäurenkommt, die durch DNA-abhängige RNA-Polymerase katalysiert wird und zur Bildung von messenger-RNA (mRNA), transfer-RNA (tRNA), ribosomaler RNA(rRNA; vgl. Abb. 1 ) und einer Reihe weiterer RNA-Spezies führt. Die Transkription ähnelt der DNA-Replikation dahingehend, daß eine matrizenabhängige Synthese von Nucleinsäuren aus Nucleosidtriphosphaten (2'-Desoxyribonucleosid-5'-triphosphate, Ribonucleosid-5'-triphosphate) erfolgt. Sie unterscheidet sich von dieser aber auch in wesentlichen Merkmalen: Bei der Transkription wird nur ein DNA-Strang (Matrizenstrang, codogener Strang) und von diesem nur ein kleiner Teil in RNA umgeschrieben. Hinzu kommt, daß RNA-Polymerasen die Fähigkeit zum sog. proof-reading (DNA-Polymerasen) fehlt. Bei der Transkription werden als energiereiche Vorstufen bzw. Substrate die 4 Ribonucleosid-5'-triphosphateATP (Adenosintriphosphat), CTP (Cytidintriphosphat), GTP (Guanosin-5'-triphosphat) und UTP (Uridin-5'-triphosphat) umgesetzt und sukzessive in Form der Ribonucleosidmonophosphate dieser Vorstufen in die vom 5'- zum 3'-Ende hin schrittweise wachsenden RNA-Ketten eingebaut. Auf diese Weise werden die Nucleotidsequenzen der Gene (DNA) in Form einzelner RNA-Ketten kopiert, d.h. von DNA zu RNA „umgeschrieben“ – daher die Bezeichnung Transkription. Die Transkription stellt den ersten Teilschritt bei der Ausprägung der in DNA verschlüsselten genetischen Information (Genexpression) dar und vollzieht sich in den 3 Phasen Initiation (Kettenstart), Elongation (Kettenverlängerung) und Termination (Kettenabbruch oder -abschluß). Vor allem für Initiation und Termination sind neben der RNA-Polymerase meist zusätzliche Proteineerforderlich. Dies sind bei Prokaryoten z.B. das cAMP bindende Protein und Sigma-Faktoren zur Initiation und der rho-Faktor (Rho-Protein) zur Termination (vgl. Abb. 2 ). Die Elongation wird z.B. durch das Nus-A-Protein beeinflußt. Bei Eukaryoten vermittelt eine Vielzahl von Transkriptionsfaktoren insbesondere die Initiation der Transkription, wobei die Bindung dieser Faktoren an die regulatorischen Sequenzen von Genen wesentlich von der lokalen Chromatinorganisation (Eu- und Hetero-Chromatin) bzw. der Anordnung der Nucleosomen sowie der DNA-Methylierung mitbestimmt wird. Regulatorische Sequenzen auf der DNA, die den Initiationspunkt und die Initiationshäufigkeit festlegen, sind die Promotoren. Durch Bindung der generellen Transkriptionsfaktoren am Promotor bildet sich der Präinitiationskomplex__; mit Bindung der RNA-Polymerase und eines Nucleosidtriphosphats spricht man vom Initiationskomplex. Die Häufigkeit der Transkription einzelner Gene bzw. Gengruppen sowie die Effizienz der Transkription wird durch eine Vielfalt von Signalstrukturen reguliert. Bei eukaryotischen und viralen Genen (Viren) kann die Transkription z.B. durch sog. enhancer verstärkt werden – DNA-Abschnitte, die an nahezu beliebigen Positionen (bis zu mehreren kb [= Kilobasen; Kilobasenpaare] vor, aber auch innerhalb oder hinter der codierenden DNA-Region) und in beliebiger Orientierung bezüglich des betreffenden Gens vorkommen können. Sog. silencer im flankierenden Bereich eukaryotischer und viraler Gene reduzieren die Häufigkeit der Initiation. Die den Kettenabschluß vermittelnden Signalstrukturen werden als Terminatoren bezeichnet (weitere Informationen zur Transkriptionskontrolle__: differentielle Genexpression, Genregulation, Response-Element; Antitermination, Attenuatorregulation, transkriptionelle Antitermination). – Die Transkription verläuft in einem Komplex aus DNA, RNA und RNA-Polymerase, den man aufgrund seiner elektronenmikroskopisch auffälligen Struktur Transkriptionsblase nennt ( vgl. Abb. 3 ). Produkte der Transkription sind einzelsträngige RNA-Ketten, die zum codogenen Strang der DNA-Matrize komplementär sind bzw. in der Nucleotidsequenz mit dem nichtcodogenen DNA-Strang identisch sind (aber: Uracil anstelle von Thymin und Ribose an Stelle von Desoxyribose). Im Rahmen der Prozessierung werden die Primärtranskripte zu den reifen, meist kürzerkettigen RNAs (mRNAs, rRNAs oder tRNAs) umgewandelt, bei sog. Mosaikgenen (Genmosaikstruktur) ist dieser Vorgang mit dem Spleißenidentisch. Eukaryotische mRNAs werden außerdem durch Capping am 5´-Ende und durch Polyadenylierung am 3´-Ende modifiziert. Bei Prokaryoten erfolgt die Transkription benachbarter Gene häufig in Form einer einzigen RNA-Kette (polycistronische mRNA), wie z.B. bei den Genen des Arabinose-Operons, des Galactose-Operons und des Lactose-Operons. Zudem findet meist gleichzeitig mit der Transkription die Translation (cotranskriptionale Translation) statt. Bei Eukaryoten ist die Transkription der nucleären Gene wegen der hier vorliegenden Kompartimentierung räumlich und zeitlich von der im Cytoplasma stattfindenden Translation getrennt. Die gebildeten Primärtranskripte (hn-RNA__) werden nach der Prozessierung im Zellkern durch den Kernporenkomplex in das Cytoplasma transportiert. In Mitochondrien findet die Transkription an beiden Strängen der mtDNA (mitochondriale DNA) statt. Die Transkripte entsprechen der kompletten Sequenz je eines der beiden mtDNA-Stränge und werden noch während der Transkription durch RNasen (Ribonucleasen) in die einzelnen tRNAs, rRNAs und mRNAs zerschnitten (mitochondriale RNA). Ähnlich verläuft die Transkription in Plastiden. Viele Gene des Plastoms sind wie bei Prokaryoten polycistronisch organisiert. Einige Gene zeigen eine für Eukaryoten typische (und auch im mitochondrialen Genom gefundene) Genmosaikstruktur, wobei Exonen weit auseinander und auf unterschiedlichen DNA-Strängen lokalisiert sein können und durch trans-Spleißen miteinander verbunden werden müssen. – Verschiedene Wirkstoffe vermögen die Transkription sehr spezifisch zu inhibieren, z.B. durch Interkalation in die doppelsträngige Matrizen-DNA (Actinomycine), durch Blockierung der RNA-Polymerase (Rifampicin bei Prokaryoten, Amatoxine bei Eukaryoten) oder durch Verhinderung der Kettenverlängerung (Cordycepin), und sind damit für analytische und therapeutische Zwecke von Interesse. – Methoden zur Analyse der Transkription bzw. der RNA-Transkripte sind u.a. in-vitro-Transkription, in-vivo-Analyse mittels Reportergenen, Northern-Technik (blotting-Techniken), Nuclear-run-on-Transkription, primer-extension-Analyse, RNase-Schutzexperimente, RT-PCR (Polymerase-Kettenreaktion), run-off-Transkription, S1-Kartierung. – Gegensatz: reverse Transkription (reverse Transkriptase). Biochemie (Geschichte der), Genaktivierung, RNA-Editing, transkriptionelle Antitermination, Transkriptionssystem, Transkriptom. –Transkription – Translation .
H.K./E.G./M.B.
Transkription
Zwischen der Transkription prokaryotischer (Protocyte) und eukaryotischer Zellen (Eucyte) bestehen, was die biochemischen Grundlagen der RNA-Synthese anbelangt, generell keine Unterschiede. Jedoch sind an der Transkription bei beiden Zelltypen verschiedene Strukturen und Proteinfaktoren auf unterschiedliche Weise wirksam. So kommt z.B. bei Eukaryoten mehr als 1 RNA-Polymerase vor, so daß die verschiedenen RNA-Spezies durch unterschiedliche Enzyme synthetisiert werden. Hinzu kommt, daß die Verpackung eukaryotischer DNA in Chromosomen die Transkription beeinflußt. Allgemein läßt sich die Transkription in 3 Teilschritte unterteilen, die Initiation („Kettenstart“), bei der es zur Wechselwirkung des zu transkribierenden Gens mit der RNA-Polymerase kommt, die Elongation („Kettenverlängerung“) als Phase der RNA-Synthese und die Termination („Kettenabbruch“), die zur Beendigung der Transkriptionsprozesse und Freigabe der RNA führt.
– Initiation: Zu Beginn der Transkription kommt es zu einer Erkennung bestimmter Sequenzmotive im Promotor eines Gens durch die RNA-Polymerase. Sie wird durch gerichtete Diffusion der RNA-Polymerase entlang der DNA-Doppelhelix möglich, wobei elektrostatische Wechselwirkungen (schwache Wechselwirkungen) sowie Interaktionen des Enzyms mit Wasserstoffdonoren und -akzeptoren der DNA auftreten. Bei Prokaryoten erfolgt die spezifische Erkennung mittels des sog. Sigma-Faktors__, bei eukaryotischen Zellen wird diese durch Transkriptionsfaktoren wie z.B. TBP (= TATA-Box bindendes Protein, TATA-Bindeprotein) vermittelt. Der Sigma-Faktor ist Bestandteil des bakteriellen RNA-Polymerase-Holoenzyms und erkennt spezifische Motive in der doppelsträngigen DNA. Bei diesen handelt es sich um Sequenzen, die als Minus-10-Box (Pribnow-Box) und Minus-35-Box bezeichnet werden, da sie 10 bzw. 35 Nucleotide stromaufwärts (upstream) vom Transkriptionsstart, der das erste Nucleotid der RNA darstellt, lokalisiert sind. Im Anschluß an diese Prozesse deckt die RNA-Polymerase einen Bereich von –50 bis +20 Basenpaaren ab. Dieser sog. geschlossene Komplex wird abschließend durch partielles Entwinden der DNA in den offenen Komplex überführt. Nach erfolgter Initiation dissoziiert der Sigma-Faktor von der RNA-Polymerase, die eine Konformationsänderung durchläuft und mit der RNA-Synthese beginnt.
– Elongation: In dieser Phase bewegt sich die RNA-Polymerase in 5'-3'-Richtung (des codierenden Strangs)an der DNA entlang, wobei es am Matrizenstrang(codogener Strang) zur RNA-Synthese in 5'-3'-Richtung kommt. Innerhalb des DNA-RNA-Proteinkomplexes, der Transkriptionsblase, kommt es vor dem 3'-Ende der entstehenden RNA zu einer Entwindung von jeweils 17 Basenpaaren der DNA, der eine Verdrillung folgt, nachdem die RNA-Polymerase einen DNA-Abschnitt abgelesen hat. Durch die wiederentstehende Doppelhelix wird die RNA aus der zunächst entstandenen DNA-RNA-Helix verdrängt.
– Termination: Die Transkription erfolgt so lange, bis die RNA-Polymerase auf eine Terminatorsequenz stößt, an der es zum Anhalten der RNA-Synthese kommt und sich die RNA und das Enzym vom Komplex lösen. Bei Prokaryoten sind 2 verschiedene Arten der Termination bekannt, die sich darin unterscheiden, ob an ihr der Rho-Faktor (Rho-Protein) beteiligt ist oder nicht. Bei der Rho-unabhängigen Termination verursacht ein GC-reicher Bereich die Entstehung einer Haarnadelstruktur (engl. hairpin loop; Haarnadelschleifen; vgl. Abb. 4 ) in der RNA, wobei angenommen wird, daß sich die Transkriptionsrate aufgrund einer langsameren Entwindung der GC-reichen Sequenz ebenfalls verlangsamt und die Bildung der Haarnadelstruktur fördert. An den GC-reichen Bereich schließt sich stets eine aus 6 Nucleotiden bestehende poly(A)-Sequenz an. Die auf Ebene der RNA vorhandene poly(U)-Sequenz bildet mit der poly(A)-Sequenz der DNA nur relativ schwache Wechselwirkungen, so daß es schließlich zu einem Lösen der RNA vom Matrizenstrang und somit zur Beendigung der Transkription kommt. Bei der Rho-abhängigen Termination bindet der Rho-Faktor zunächst an die entstehende RNA und bewegt sich durch ATP-Hydrolyse (Adenosintriphosphat) auf der RNA entlang zur Polymerase, wo es zur Entwindung der DNA-RNA-Helix und letztlich zur Auflösung der Transkriptionsblase kommt.
Bei Eukaryoten erfolgt die Termination der Transkription analog zur faktorenunabhängigen Termination. Im Unterschied zu detaillierten Kenntnissen über die Struktur eukaryotischer Promotoren und Transkriptionsfaktoren liegen über eukaryotische Terminatoren weniger Informationen vor.
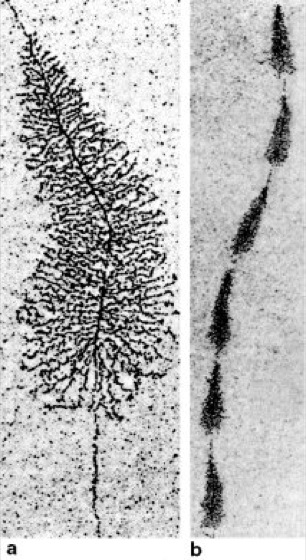
Transkription
Abb. 1: Die elektronenmikroskopischen Aufnahmen zeigen einen Transkriptionsvorgang: die Synthese von ribosomaler RNA (rRNA) entlang einem Gen für ribosomale RNA. Viele RNA-Polymerasen hintereinander synthetisieren die rRNA entlang der zentralen DNA vom Startsignal (in Abb. a oben) bis zum Stopsignal (in Abb. a unten), wo die RNA-Moleküle ihre volle Länge erreichen und freigesetzt werden (a, stärkere Vergrößerung). Bei vielen Organismen enthält die DNA gleich eine ganze Serie von Genen für die rRNA (b, geringere Vergrößerung).
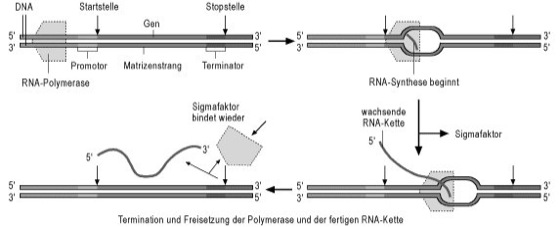
Transkription
Abb. 2: Schema der Transkription bei Prokaryoten
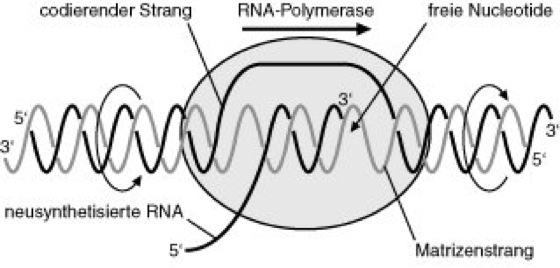
Transkription
Abb. 3: Der Komplex aus DNA, RNA und RNA-Polymerase bildet die Transkriptionsblase. Im vorderen Bereich der Transkriptionsblase wird die DNA-Doppelhelix entwunden und aufgebrochen, im hinteren Bereich wird die neusynthetisierte RNA aus der Bindung an den Matrizenstrang verdrängt und die DNA-Doppelhelix rückgebildet. Dazwischen bildet sich ein RNA/DNA-Hybrid, wobei die RNA-Polymerase die Basenpaarung des Matrizenstrang-Codons mit dem eintretenden Ribonucleotid kontrolliert und die Phosphodiesterbindung knüpft.

Transkription
Abb. 4: Haarnadelförmige Sekundärstruktur im 3'-nichtcodierenden Bereich einer prokaryotischen mRNA. Links ist ein Stopcodon für die Translation (UAA) zu erkennen, rechts im Anschluß an den GC-reichen Bereich der Haarnadelstruktur eine poly(U)-Sequenz. Diese Sequenz bildet mit der poly(A)-Sequenz der DNA relativ schwache Wechselwirkungen, was das Lösen der RNA vom Matrizenstrang bewirkt.
Genregulation
[Genregulation][Genregulation] w [von latein. regulare = regeln], genetische Regulation, Regulation der Genaktivität, Regulation der Transkription, die von dem biochemischen und biophysikalischen Zustand einer Zelle oder eines vielzelligen Organismus sowie von Umwelteinflüssen abhängige Regulation der Transkription von Genen (Gen). Die Genregulation ist von entscheidender Bedeutung für die differentielle Genexpression. Es werden 2 grundlegende Mechanismen der Genregulation unterschieden:
1) Negative Genregulation: die Bindung eines Repressors an die Kontrollregion eines Strukturgens verhindert die Transkription des Gens. Die von Regulatorgenen codierten Repressorproteine vermitteln die Signalwirkung von kleineren Molekülen, den Effektoren (Substrate oder Endprodukte biochemischer Reaktionsketten), auf die Aktivität bestimmter Gene oder Gengruppen. Hierfür ist entscheidend, daß Repressoren Spezifität zur Bindung der entsprechenden Effektoren einerseits und zur Bindung an die entsprechenden DNA-Kontrollregionen (Operatoren) andererseits besitzen. 2 Typen von Repressoren werden unterschieden: a) Repressoren, welche die Transkription von Genen eines katabolischen Stoffwechselweges (Abbau, Dissimilation, Stoffwechsel) kontrollieren, können an ihre Operatorregion nur in Abwesenheit des Effektors (Substrat eines der codierten Enzyme) anlagern und damit die Aktivität des betreffenden Gens oder Operons blockieren; b) Repressoren, welche die Transkription von Genen eines anabolischen Stoffwechselweges kontrollieren (Anabolismus), werden erst durch die Anwesenheit des Effektors (Endprodukt des Stoffwechselweges) allosterisch (Allosterie) so verändert, daß sie an die Operatorregion binden und damit den Transkriptionsprozeß blockieren. Das Prinzip der negativen Genregulation wurde 1961 von F. Jacob und J.L. Monod (Jacob-Monod-Modell) für die Regulation der Synthese zuckerabbauender Enzyme (Lactose-Operon) von Mikroorganismen postuliert. Mittlerweile sind zahlreiche andere Operonen in Prokaryoten in molekularen Details untersucht worden, deren Transkription nach demselben Prinzip reguliert wird (Arabinose-Operon, Galactose-Operon). Ein anderer Mechanismus der negativen Genregulation ist die stringente Kontrolle bei der Transkription prokaryotischer r-RNA-Gene und t-RNA-Gene (transfer-RNA). Steigt bei Mangel an Aminosäuren die Konzentration von unbeladenen t-RNAs im Cytoplasma, so wird das Nucleotid Guanosin-5'-diphosphat-3'-diphosphat (ppGpp) gebildet, das die Initiation der Transkription von r-RNA- und t-RNA-Genen inhibiert. – Die Bedeutung der negativen Genregulation bei der Steuerung der Genaktivität in eukaryotischen Zellen (Eucyte) ist vermutlich geringer als bei Prokaryoten (Protocyte). Hauptgrund dafür dürfte das im Vergleich zu Prokaryoten riesige Genom sein, von dem je nach Organismus jedoch nur ca. 7% transkribiert werden. Daraus folgt, daß der größte Teil des eukaryotischen Genoms transkriptionell inaktiviert werden muß. Dafür gibt es in Eukaryoten verschiedene Mechanismen, wie das Verpacken von nicht transkribierten Teilen des Genoms in inaktiven Chromatindomänen (Chromatin). Methylierte DNA-Abschnitte (DNA-Methylierung) werden nicht transkribiert. Gene, die in einem Gewebe hochmethyliert und inaktiv sind, können in einem anderen Gewebe nichtmethyliert vorliegen und exprimiert werden. Sogenannte silencer wirken selektiv über spezifische Protein-DNA-Interaktionen auf einzelne Gene und stellen den am schnellsten reversiblen und am ehesten mit der prokaryotischen Repression vergleichbaren Mechanismus dar. Mittlerweile kennt man auch Repressoren der Transkription, die direkt in die Ausbildung von Präinitiationskomplexen eingreifen, z.B. in Form von Protein-Protein-Interaktionen von Repressormolekülen mit Komponenten der generellen Transkriptionsmaschinerie oder mit Aktivatoren der Transkription (z.B. die Proteine Krüppel, Engrailed oder WT1).
2) Positive Genregulation: die Transkription von prokaryotischen Genen oder Gengruppen wird durch die Anlagerung von Aktivatorproteinen (wiederum Produkte von Regulatorgenen) an die Kontrollregion stimuliert. Ohne angelagerte Aktivatoren besitzt RNA-Polymerase nur geringe Affinität zu den Promotoren der entsprechenden Gene bzw. Operonen. Die Wirksamkeit des Aktivators hängt (analog zu den Repressoren) von der Gegenwart bestimmter, als Effektoren wirkender Kleinmoleküle ab. Ein Beispiel positiver Genregulation bei Escherichia coli stellt die Katabolitrepression dar: bei Abwesenheit von Glucose im Medium steigt die intrazelluläre Konzentration von cAMP (zyklisches Adenosinmonophosphat), wodurch die Transkription mehrerer Operonen, welche die Enzyme zur Vergärung anderer Zucker codieren, stimuliert wird. Dabei bindet cAMP, das Effektormolekül, an das cAMP bindende Protein, das als Aktivatorprotein dieser Operonen wirkt (Arabinose-Operon, Galactose-Operon, Lactose-Operon). Die positive Genregulation ist in diesem Fall der spezifischen negativen Genregulation jedes einzelnen Operons übergeordnet. – Bei Eukaryoten ist die positive Transkriptionskontrolle das dominierende Prinzip der Genregulation. Die Regulation der Transkriptionseffizienz erfolgt im wesentlichen durch Bindung von spezifischen Transkriptionsfaktoren an Kontrollregionen (TATA-Box, CCAAT-Box; enhancer) auf der DNA, die im Fall der RNA-Polymerase-I- und -II-transkribierten Gene im 5'-flankierenden Bereich der Gene liegen. Solche Protein-DNA-Interaktionen sind Voraussetzung für die Transkriptions-Initiation. Die verschiedenen Genklassen eukaryotischer Zellen werden durch 3 unterschiedliche RNA-Polymerasen transkribiert, die durch unterschiedliche generelle Transkriptionsfaktoren (GTF) unterstüzt werden (RNA-Polymerase I, II und III entsprechend durch TFI, II und III [Transkriptionsfaktoren] sowie TAFI, II und III [TBP-assoziierte Faktoren]). Alle 3 RNA-Polymerasen nutzen zusätzlich den gemeinsamen generellen Transkriptionsfaktor TBP (TATA-Bindeprotein). Die „Promotorstärke“, d.h. das Potential eines Promotors, die Transkription zu aktivieren, hängt von der genauen Nucleotidsequenz des Promotorbereichs ab, da dies die Affinität der Transkriptionsfaktoren für diese DNA-Region steuert. Durch unterschiedliche „Promotorstärken“ werden verschiedene Gene unterschiedlich stark transkribiert. Die Spezifität der Transkriptionsinitiation wird durch das Zusammenwirken von verschiedenen Transkriptionsfaktoren erreicht. Es ergibt sich eine basale Transkriptionsmaschinerie (sog. Präinitiationskomplex) mit beeindruckender Komplexizität aus vielen Proteinkomponenten, wobei die generellen Transkriptionsfaktoren (GTF) selbst aus mehreren Untereinheiten bestehen. Die differentielle Genaktivität, d.h. das Muster der eukaryotischen Transkription in Abhängigkeit von zeitlichen, räumlichen, zelltypspezifischen, organspezifischen oder signalvermittelten Parametern, wird wesentlich durch die Aktivität regulatorischer Transkriptionsfaktoren (RTF) definiert, die direkt oder indirekt mit den GTF der basalen Transkriptionsmaschinerie in Interaktion treten und zur Aktivierung beitragen. Bei einer gewebe- oder stadienspezifischen Genaktivierung spielen besonders die enhancer-bindenden Proteinfaktoren, deren Erkennungssequenz sowohl upstream als auch downstream vom transkribierten Bereich (oder sogar in einem Intron) liegen kann, eine entscheidende Rolle. Über derartige regulatorisch wirksame Proteinfaktoren nehmen auch extrazelluläre Faktoren, u.a. Licht, Temperatur (heat-shock-Proteine) und Wachstumsfaktoren, Einfluß auf die Genregulation (Signaltransduktion). Die Transkriptionsfaktoren weisen einige charakteristische DNA-Bindemotive auf (Protein-DNA-Interaktion), die spezifische Sequenzen, oft gekennzeichnet durch palindromische Sequenzsymmetrie (Palindrom), auf der DNA erkennen. Voraussetzung für die Bindung von spezifischen Proteinfaktoren an DNA-Kontrollelemente kann eine lokale Auflösung der Struktur der Nucleosomen sein (DNase I-hypersensitive Stellen). Auf der Chromatinstruktur beruhende Parameter, z.B. das Nucleosom mit seinem Histonkern oder HMG-Proteine, können potentiell reprimierende oder auch aktivierende Wirkung auf die Genaktivität haben. Bespielsweise kann die topologische Faltung der in Nucleosomen verpackten DNA zu gewünschten benachbarten Positionierungen von ansonsten weit entfernt liegenden DNA-Bereichen führen. Hilfsproteine (SWI/SNF-Komplexe) vermitteln dynamische Chromatinveränderungen zur Erleichterung der Transkription oder deren Regulation. Das Ausmaß der Transkriptionsinitiation kann in seltenen Fällen auch durch DNA-Translokationen beeinflußt werden. Dabei kann es geschehen, daß ein Gen unter die Kontrolle stärkerer Transkriptionsstartsignale gerät und deshalb mit einer erhöhten Rate exprimiert wird (Aktivierung von Onkogenen). Derartige Mechanismen werden mit der Entstehung bestimmter Tumoren in Zusammenhang gebracht.
In Hefe werden die Gene für die beiden unterschiedlichen Paarungstypen a und α in vegetativ reproduzierenden Zellen nicht exprimiert. Unter bestimmten Umständen werden jedoch entweder die a- oder die α-Typ-Gene in den MAT-Locus verschoben. Nur in dieser Position werden sie exprimiert. Das Produkt des MAT-Locus der Hefe – ebenso wie die Homöobox-Sequenz (Homöobox) von Drosophila melanogaster und homologe Sequenzen bei anderen Organismen – scheinen als Regulationselemente für zerstreut liegende Gene zu fungieren, die im Verlauf der Entwicklung simultan aktiviert werden. Es wird angenommen, daß die Gene, die durch die Produkte homöotischer Gene induziert werden, Sequenzen aufweisen, die mit bakteriellen Promotoren oder möglicherweise viralen enhancern verglichen werden können und an die die regulatorischen Proteine, die die Transkription stimulieren, binden. In vielen eukaryotischen Zelltypen existieren Mechanismen zum Transponieren von Genen auf einem Chromosom. Die Umordnung von Genen, wie sie bei den Hefe-Paarungstyp-Genen vorkommen, ist für die Immunglobulin- und T-Zellen-Antigen-Rezeptormoleküle ein notwendiger Schritt zur Aktivierung der entsprechenden Lymphoidklone.
Die Aktivierung spezifischer Transkriptionsfaktoren wird meist durch Mechanismen der Signaltransduktion ausgelöst. So beruht die Signalwirkung der meisten Hormone, Cytokine und anderer Substanzen auf deren Bindung an plasmamembranständige Rezeptoren und im folgenden phosphorylierten cytoplasmatischen Substratproteinen, die entweder selbst als Transkriptionsfaktoren aktiv werden oder solche Faktoren aktivieren. – Neben der Regulation der Transkriptionsinitiation kann auch die Steuerung einer vorzeitigen Termination, wie sie bei der Attenuatorregulation vorliegt, zur Genregulation beitragen. Außerdem kann auch die helikale Torsion der DNA (DNA-Topoisomerasen) die Effizienz der Transkription beeinflussen. Die Kontrolle der Synthese der messenger-RNA stellt zwar den ersten und häufig entscheidenden Schritt in der Steuerung der Genaktivität dar, aber auch nach erfolgter Transkription gibt es noch weitere Möglichkeiten, die Genexpression zu regulieren. So kann z.B. über m-RNA-Stabilität, Translationskontrolle und Proteinstabilität Einfluß auf die Genwirkung genommen werden. – Störungen der Genregulation, z.B. durch defekte Transkriptionsfaktoren, können u.a. Entwicklungsstörungen, hormonelle Störungen, Krebserkrankungen sowie Syndrome defekter DNA-Reparatur hervorrufen. Genregulation , Gentechnologie .
